
차세대 염기서열 분석 (NGS(Next Generation sequencing; NGS) 기법의 발달로 유전체 연구가 급격히 발전하면서, 다양한 시퀀싱 기법들이 개발되었습니다.많은 샘플의 유전체를 서로 비교하기 위해 Reduced representation genome sequencing (RRGS)이라는 기술이 개발되었습니다. 이름에서 알 수 있듯 유전체 일부 지역에 대한 효율적인 스크리닝을 통해 ‘해당 영역이 유전체를 축소(reduced)하여 대변한다(representation)’ 라는 컨셉을 가지고 있습니다.
이를 통해 수십~수백 개 샘플의 유전체 내 변이를 파악하고, 해당 영역에 대한 유전 정보까지 파악할 수 있습니다.
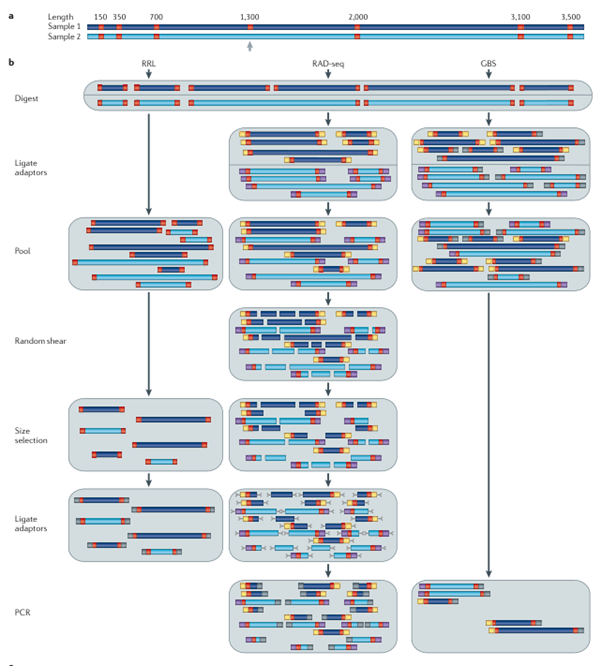
이 기술을 이용한 시퀀싱 생산 방법 중 가장 유명한 것이 Genotyping-by-sequencing (GBS) 과 Restriction site-associated DNA sequencing (RAD-seq) 기법입니다.
특히 이번 글에선 RAD-seq에 대해 소개하고자 합니다.
RAD Sequencing:
RAD sequencing 기법은 Whole genome sequencing (WGS)와 같은 유전체 분석 방식이지만, 유전체 전체를 시퀀싱하는 WGS와 달리, 달리 제한효소(Restriction enzyme)를 이용하여 유전체를 절단하여 만든 DNA fragment를 이용해 염기서열을 분석하는 기법입니다.
제한효소 인식 부위(restriction site) 주변 서열만 부분적으로 해독하면서, 유전체 전체에 걸쳐 정보를 확보할 수 있고 시간과 비용이 절감된다는 장점이 있어 다양한 종간 비교 분석이나 집단유전분석, 계통분석, 유전지도 작성 등 Genotyping 연구에서 점차 많이 사용되고 있습니다.


GBS와의 비교:
GBS 또한 제한효소(Restriction enzyme)를 이용하여 유전체를 절단하여 만든 DNA fragment를 Library 제작 시 insert fragment로 삽입하여 제작하게 됩니다.
단, sonicator를 이용한 Random shearing 과정이 생략되고, 따로 DNA fragment selection 과정을 거치지 않는다는 차이가 있습니다.
따라서 어떤 DNA fragment가 길다면 시퀀싱되는 어댑터 부근 말단 부위가 서로 멀어 중간 부분은 시퀀싱되지 않는 경향이 있을 수 있습니다.
이러한 부분은 적정 restriction enzyme을 선택하거나, GBS library QC 시에 library의 크기가 적절한지를 파악하여 보완합니다.
RAD Sequencing의 파생 기술들:
RAD-seq은 라이브러리 제작 과정에서 차이가 나는 여러 파생 기술을 가지고 있으며, 연구 목적과 상황에 따라 다르게 활용됩니다.

대표적인 기술 몇 가지를 소개해드립니다.
- Original RAD seq: 가장 기본적인 RAD sequencing 기술로, 하나의 제한효소를 사용하여 유전체를 절단합니다. Locus 수를 조절할 수 있는 옵션이 많고, 대규모/복잡한 유전체에도 적용 가능하지만 비용과 노력이 많이 필요합니다.
- 2bRAD seq.: 타입 IIB 제한효소를 사용하여 33-36bp의 일정한 길이로 DNA 절편을 생산하는 기술입니다. 비용이 상대적으로 덜 들어가나, Locus 수를 조절할 수 있는 옵션이 제한적이고 대규모/복잡한 유전체에는 적용하기 어렵다는 단점이 존재합니다.
- GBS (Genotyping by Sequencing): RADseq과 마찬가지로, 하나의 제한효소를 사용하여 유전체의 일부분을 시퀀싱합니다. RADseq에 비해 실험 절차를 줄여 비용이 적다는 장점이 있으나 random shearing 방식이라 라이브러리의 정확한 사이즈 선정은 어렵습니다.
2RAD/3RAD seq: 두/세 가지 제한효소를 사용하여 유전체를 양끝이 서로 다른 제한효소로 잘린 DNA 절편으로 더욱 정밀하게 절단하고 분석하는 기술입니다. Locus 수 조절이 가능하고 대규모 유전체에도 적합한 반면 상대적으로 비용은 낮은 편입니다.
3RAD 기술의 장점:
3RAD (triple enzyme RAD)는 세 가지 제한효소를 사용하여 양끝이 서로 다른 제한효소 인식 부위를 갖도록 유전체를 절단하는 기술입니다.
세 가지 제한효소 중 두 가지 제한효소는 제한효소 절단 후 점착성 말단(sticky end) 서열이 동일한 제한효소를 사용하여 절단합니다.
이를 통해 NGS 어뎁터(adapter) 간의 이량체(dimer) 형성을 최소화하여 어뎁터 결찰(adapter ligation) 효율이 증가하며, 한번의 시퀀싱을 통해 얻은 염기서열의 판독할 수 있는 범위 (coverage)가 늘어나는 장점이 있습니다.
이러한 점을 통해 3RAD는 기존의 장점인 재현성, 확장성에 더해 유연성까지 제공합니다.
- 재현성: 다양한 제한효소를 사용하여 일관된 절단 패턴을 생성함으로써 실험 결과의 재현성을 높일 수 있습니다.
- 확장성: 여러 제한효소를 사용함으로써 다양한 비모델 생물에서도 많은 변이를 탐지할 수 있어 참조 유전체 없이도 높은 해상도의 유전체 지도를 작성할 수 있습니다.
- 유연성: 특정 연구 목적에 맞추어 다양한 조합의 제한효소를 사용할 수 있어 연구의 유연성을 제공합니다.
논문 사례
몇 가지 논문 사례 소개해드리고 마무리하고자 합니다.

Are Genomic Updates of Well-Studied Species Worth the Investment for Conservation? A Case Study of the Critically Endangered Mag
Abstract. As genomic-scale data sets become economically feasible for most organisms, a key question for conservation biology is whether the increased reso
academic.oup.com
멸종위기종 Magdalena river turtle의 과거 분자 마커 연구를 재검토하기 위해 3RAD 기법으로 집단 유전 분석을 수행한 논문으로 중립 유전 마커의 수를 수천 배로 증가시켜 개체군 구조와 거리별 격리 여부를 확인한 사례입니다.

Comparative Analysis of SNP Discovery and Genotyping in Fagus sylvatica L. and Quercus robur L. Using RADseq, GBS, and ddRAD Met
Next-generation sequencing of reduced representation genomic libraries (RRL) is capable of providing large numbers of genetic markers for population genetic studies at relatively low costs. However, one major concern of these types of markers is the precis
www.mdpi.com
같은 종 샘플의 DNA 서열을 RRGS 방법인 GBS, RADseq, ddRADseq 기법을 이용해 시퀀싱하고, 각 기법별로 데이터를 비교한 논문 사례입니다.
결론:
RAD sequencing과 그 파생 기술들은 유전체 연구에서 중요한 역할을 하며, 다양한 생물학적 질문에 답을 찾기 위한 강력한 도구입니다.
특히 3RAD와 같은 최신 기술들은 고해상도 유전체 분석을 가능하게 하여, 더 정밀한 유전체 변이 탐지와 유전자 기능 연구를 가능하게 합니다.
이러한 기술들의 발전은 유전체 연구의 새로운 가능성을 열어주며, 앞으로의 연구에 큰 기여를 할 것으로 기대됩니다.
3RAD 기술에 대한 더 자세한 이야기와 최첨단 데이터 분석 솔루션에 대해서는 씨더스로 문의 바랍니다!
▼ 씨더스 홈페이지 바로가기
참고 문헌
- Miller, M. R., et al. (2007). Rapid and cost-effective polymorphism identification and genotyping using restriction site associated DNA (RAD) marker. Genome research.
- Wang, S., et al. (2012). 2b-RAD: a simple and flexible method for genome-wide genotyping. Nature Methods.
- Andrews, K. R., et al. (2016). Harnessing the power of RADseq for ecological and evolutionary genomics. Nature review.
- Bayona-Vaquez, N. J. et al. (2019). Adapterama Ⅲ: Quadruple-indexed, double/triple-enzyme RADseq libraries (2RAD/3RAD). PeerJ 7.
'SEED DB' 카테고리의 다른 글
| 씨더스, 농업에 AI 기술을 스마트하게 접목하다 (9) | 2024.10.04 |
|---|---|
| 한국형 스마트팜의 세대별 특징 (0) | 2024.08.26 |
| 분자마커 시리즈 1편: 분자마커에 대한 이해 (3) | 2024.07.25 |
| 식물 표현체 분석 기술 시리즈 1편: 2D 이미지 분석 (12) | 2024.07.22 |
| 표현형 이해하기: 종합 가이드 (7) | 2024.07.15 |




